新冠病毒(SARS-CoV-2)在全球范围内大肆传播,新冠变异株的流行使疫情防控更具挑战。快速筛查是控制疫情传播最有效的手段。RT-qPCR是目前新冠病毒的检测金标准。该方法特异性强、灵敏度高, 但是由于其依赖实验室专业仪器和专业人员操作,存在耗时长等问题(从采样到出结果大致需要6小时),难以应对病毒惊人的传播速度。因此,亟须研发一种具备高灵敏度、高特异性、操作便捷且快速的核酸检测方法。
2022年3月21日,武汉大学医学研究院、中南医院医学研究院、教育部免疫与代谢前沿科学中心殷昊教授和张楹教授课题组合作在Nature Biomedical Engineering上发表了题为 Fast and sensitive detection of SARS-CoV-2 RNA using suboptimal protospacer adjacent motifs for Cas12a的研究论文。研究者首次靶向非经典PAM建立了兼具快速(15-20分钟)、灵敏(和qPCR相当)、稳定和特异的核酸检测一步法(sPAMC),并在SARS-CoV-2真实样本中达到94.2%准确度并无一例假阳性。sPAMC技术仅需20分钟就能检测到Ct值为35.8的新冠真实样品,并且用便携式紫外灯或蓝光灯照射即可观察到结果。

CRISPR/Cas系统不仅用于基因编辑,也被研发为核酸检测工具。Cas12和Cas13特异性切割双链DNA底物(顺式切割)后,会激活其非特异性切割单链DNA或RNA的能力(反式切割)1-5。CRISPR特异性由两部分共同决定,一部分是crRNA和靶DNA之间的碱基配对,另一部分是Cas蛋白复合体和位于靶DNA旁的短序列形成非共价结合,后者被称为PAM (Protospacer Adjacent Motif)。当Cas12搜寻到底物上的PAM序列,在crRNA和靶DNA碱基配对之后,进而特异性切割dsDNA,随后激活其反式切割能力,快速降解单链DNA报告系统,释放出的荧光信号。新冠病毒检测的两步法(SHERLOCK和DETECTR)先通过等温扩增富集待检的核酸片段,再加入CRISPR/Cas切割体系产生信号。虽然两步法兼具高特异性、高灵敏度等优点,却增加了操作复杂度和引入交叉污染等问题6,7。STOPCovid是在两步法基础上优化出的一步检测法,即在一个反应试管中同时进行样品扩增和切割反应,虽然简化了操作步骤并降低交叉污染风险,但灵敏度显著低于两步法,且需要约45分钟至1小时左右的反应时间8,9。
研究者们致力于研发能满足快速简便、灵敏特异的一步法核酸检测新方法。通过比较新冠病毒序列中四个位点的非经典PAM (VTTV,TVTV, VTTV) 和经典PAM (TTTV) 的反式切割活力和一步法效率,发现和经典PAM相比,非经典PAM显著加快检测速度,将灵敏度提高了10-100倍,大幅度提升信号的稳定性。进一步探索机制发现:在一步法反应中,等温扩增和CRISPR检测存在竞争关系。当Cas12a遇到经典PAM时,由于切割能力过强,等温反应扩增出来的目的片段被快速消耗,难以达到指数扩增,导致底物无法有效富集,荧光信号延迟产生且不稳定;而对于非经典PAM介导的一步法,Cas12a和底物的结合能力弱。在反应初期,等温扩增占据主导地位,快速富集足够的底物,为反应后期Cas12a切割和荧光释放奠定前期基础。
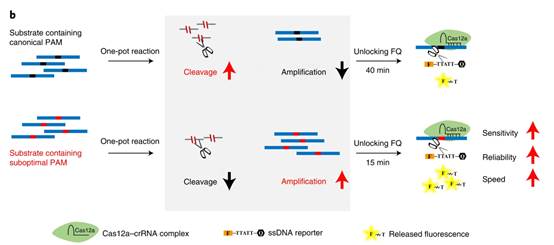
图1. 非经典和经典PAM介导的一步法快检方法概览图
研究者们将非经典PAM介导的一步法检测技术命名为sPAMC (for suboptimal PAM of Cas12a-based test with enhanced flexibility, speed, sensitivity, and reproducibility),并将sPAMC应用到SARS-CoV-2真实样品检测。研究者们共检测了204个咽拭子样本,在104个RT-qPCR阳性样本中,sPAMC 能检出98个,剩余的100个阴性样本均未检出,证明了sPAMC 有94.2%的检出率且无假阳性。sPAMC技术仅需20分钟就能检测到Ct值为35.8的新冠真实样品,并且仅用便携式紫外灯或蓝光灯照射即可观察到结果。
非经典PAM的快检方法sPAMC 相比于传统一步法有如下优点:1)检测速度提高2-3倍,可以实现在10-15分钟内检测出DNA病毒,15-20分钟内检测出RNA病毒;2)荧光信号结果在样本间的可重复性高,荧光信号波动少于30%;3)检测灵敏度提高,与qPCR一致; 4) 极大地拓宽了靶向的检测位点可选择性,可选择的非经典PAM组合为经典PAM的7倍以上。研究者首次提出了通过靶向非经典PAM,降低Cas12a的顺式和反式切割速率的方法来实现核酸检测,为开发建立更快更灵敏的CRISPR核酸检测方法提供了新思路。
本研究获得武汉大学医学研究院仪器设备共享中心、武汉大学中南医院和中国医学科学院武汉感染性疾病及肿瘤研究中心的支持。本项工作得到了国家自然基金和科技部重点研发计划的资助。
原文链接:
https://www.nature.com/articles/s41551-022-00861-x
1. Chen, J. S. et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science 360, 436-439 (2018).
2. Teng, F. et al. CDetection: CRISPR-Cas12b-based DNA detection with sub-attomolar sensitivity and single-base specificity. Genome biology 20, 1-7 (2019).
3. East-Seletsky, A. et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection. Nature 538, 270-273 (2016).
4. Gootenberg, J. S. et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science 356, 438-442 (2017).
5. Myhrvold, C. et al. Field-deployable viral diagnostics using CRISPR-Cas13. Science 360, 444-448 (2018).
6. Broughton, J. P. et al. CRISPR–Cas12-based detection of SARS-CoV-2. Nature biotechnology 38, 870-874 (2020).
7. Patchsung, M. et al. Clinical validation of a Cas13-based assay for the detection of SARS-CoV-2 RNA. Nature biomedical engineering 4, 1140-1149 (2020).
8. Joung, J. et al. Detection of SARS-CoV-2 with SHERLOCK one-pot testing. New England Journal of Medicine 383, 1492-1494 (2020).
9. Arizti-Sanz, J. et al. Streamlined inactivation, amplification, and Cas13-based detection of SARS-CoV-2. Nature communications 11, 1-9 (2020).