CRISPR/Cas行使编辑功能依赖于识别靶标DNA上的特殊序列(简称PAM)。PAM序列的复杂程度决定了可编辑位点的上限。但在实际应用中,常常受限于编辑位点没有PAM序列,导致无法进行有效编辑。已知的Cas蛋白主要通过和PAM序列形成多个氢键,进而弯曲并打开双链DNA,最终切割DNA (Sternberg et al., 2014)。DNA在真核细胞里具有不同的拓扑结构,比如经典的右手螺旋B-form和特殊的左手螺旋Z-form。转录过程中,因为打开DNA双链引起扭力变化,导致转录上游和转录下游的DNA分别形成负超螺旋和正超螺旋构象。研究表明,因不同拓扑结构的DNA其扭力不同,导致打开双链难易程度也不同(Stolz et al., 2019)。然而,DNA拓扑结构是否可以在调节PAM识别和结合中发挥作用仍然未知。
2022年10月21日,武汉大学医学研究院、免疫与代谢前沿科学中心和武汉大学中南医院的张楹团队在Molecular Cell杂志上发表了题为DNA topology regulates PAM-Cas9 interaction and DNA unwinding to enable near PAMless cleavage by thermophilic Cas9的论文。报道了来源于嗜热菌、天然具有广谱PAM且高活力的AtCas9。AtCas9的PAM为N4CNNN和N4RNNA (R=A或G), 可以覆盖68%的基因组序列。研究揭示其和PAM互作的新机制,发现除了经典氢键互作之外,AtCas9的特殊loop结构可嵌入DNA大沟,且其嵌合度受DNA构象调控,实现对特定DNA构象进行不依赖PAM的任意切割。此外,虽然AtCas9的最适酶活温度是55℃,AtCas9可在哺乳动物细胞中表现出高效的碱基编辑活力,在多个位点达到60%的编辑效率。GUIDE-seq脱靶分析显示,相比于SpCas9, AtCas9并没有明显的脱靶现象。论文首次报道天然具有广谱PAM的新型基因编辑工具,且可在细菌中不依赖PAM序列切割质粒,在真核细胞具备高效编辑活力。
首先,作者通过阳性筛选和阴性筛选两个方法分别对AtCas9的PAM进行鉴定,发现当底物是负超螺旋(质粒)构象时,没有任何序列偏好性;当底物是线性构象时(B-form),在第5位有强烈的C/A/G序列富集(图1)。当将PAM的第5位C突变为T时,AtCas9完全丧失了对线性DNA的切割但却不影响其对松散构象DNA(负超螺旋和Z-form)的切割活性。另外,将AtCas9表达系统构建到大肠杆菌中,并转入16种包含PAM或非PAM的质粒构象底物时, AtCas9对质粒底物展现出不依赖PAM的切割活性。
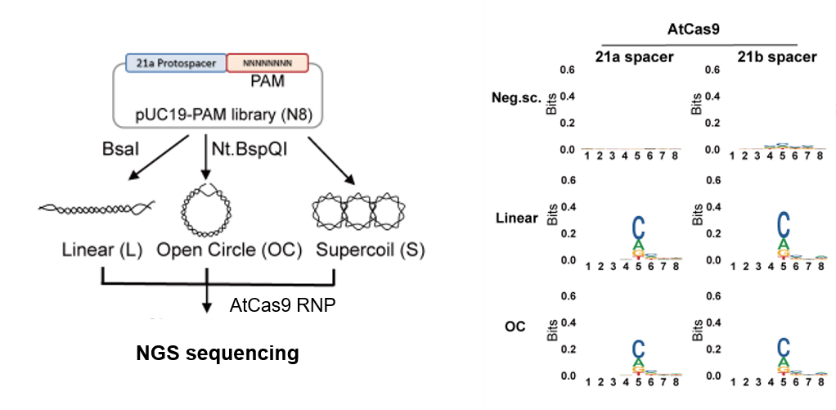
图1.PAM偏好性。当底物是质粒构象时,没有序列偏好性;当底物是线性或开环构象时,第五位有C/A/G的富集。
为了剖析DNA拓扑结构如何影响PAM识别,作者探究了拓扑结构在调节Cas9结合中的作用。不同于SpCas9,AtCas9在RNP高浓度下仍可结合PAM突变的线性DNA。为了直接验证DNA构象可以直接影响PAM-Cas9的互作,作者利用不含有protospacer的多个串联突变PAM底物进行竞争实验。结果表明,松散构象的突变PAM串联体可高效竞争含有PAM的完美底物,提示DNA拓扑结构可直接影响AtCas9蛋白和PAM的互作。为了揭示PAM-AtCas9相互作用机制,作者利用AlphaFold2和SWISS模型分别预测AtCas9的结构,发现AtCas9蛋白有一特殊loop结构域,可紧密地嵌入DNA大沟中,作为支撑Cas9-DNA互作的锚点。同时,loop结构中Asp1089可以与PAM上C5形成氢键,提供了序列特异性。这些结果表明,独特的AtCas9-PAM相互作用模式和 DNA 拓扑结构共同决定了 AtCas9 最终对底物的切割活性。
为了验证嗜热AtCas9在哺乳动物细胞中是否有活性,研究者对AtCas9进行一系列改进,在EGFP位点中达到60%的切割效率。利用GUIDE-Seq检测AtCas9的脱靶效应,发现AtCas9的脱靶事件远低于SpRY(一种近乎无PAM的SpCas9变体)。考虑到 PAM 选择对碱基编辑器的高度限制,作者进一步探索了AtCas9在碱基编辑中的应用。AtCas9-BE3可以对超螺旋质粒底物进行C到T的单碱基编辑,在含有PAM (CNNN和RNNA)的底物中平均编辑效率达到46%。在内源性基因位点中,AtCas9-BE3可产生与SpRY-BE3相当的单碱基编辑效率,平均编辑效率为30.91%。同样的,AtCas9-ABE8e也可在内源性基因位点发挥功能,平均编辑效率为16.72%。
小结:作者发现了天然具有广谱PAM的新型Cas9(AtCas9),且DNA拓扑结构可调节Cas9与PAM的识别。AtCas9的PAM识别结构域中的loop motif可作为额外的锚定点启动DNA解旋,其中Asp1089既可与PAM第5位C发生氢键相互作用,又与T存在空间排斥力,揭示了一种全新Cas9-PAM相互作用机制。另外,AtCas9能够介导哺乳动物基因组中的有效碱基转换,其对松散构象的广谱 PAM 编辑为基因组编辑家族增加了一个新工具。
武汉大学医学研究院研究生史亚晶、段敏以及云南师范大学丁俊美为本文共同第一作者,武汉大学医学研究院张楹教授为唯一通讯作者。感谢马赛诸塞医学院Erik Sontheimer教授、武汉大学殷昊教授、普颖颖教授对本项研究提供帮助。
论文全文链接:https://www.sciencedirect.com/science/article/pii/S1097276522009534?via%3Dihub
参考文献
Sternberg, S.H., Redding, S., Jinek, M., Greene, E.C., and Doudna, J.A. (2014). DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature 507, 62-67. 10.1038/nature13011.
Stolz, R., Sulthana, S., Hartono, S.R., Malig, M., Benham, C.J., and Chedin, F. (2019). Interplay between DNA sequence and negative superhelicity drives R-loop structures. Proc Natl Acad Sci U S A 116, 6260-6269. 10.1073/pnas.1819476116.

水中洛神起舞:翩若惊鸿,婉若游龙
洛神比拟AtCas9, 琵琶比拟PAM,彩带代表双链DNA。当彩带紧密缠绕时,洛神需借助琵琶在彩带中翩翩起舞;当彩带松散缠绕时,洛神自由飞舞跳跃于彩带之中。